LA RESISTENCIA A ANTIBIÓTICOS SE PROPAGA EN UNO DE LOS PATÓGENOS MÁS PELIGROSOS

• El estudio, publicado en la revista Nature Communications, es coliderado por Adrián Cázares López, exbecario postdoctoral de la Secretaría de Educación, Ciencia, Tecnología e Innovación (SECTEI)
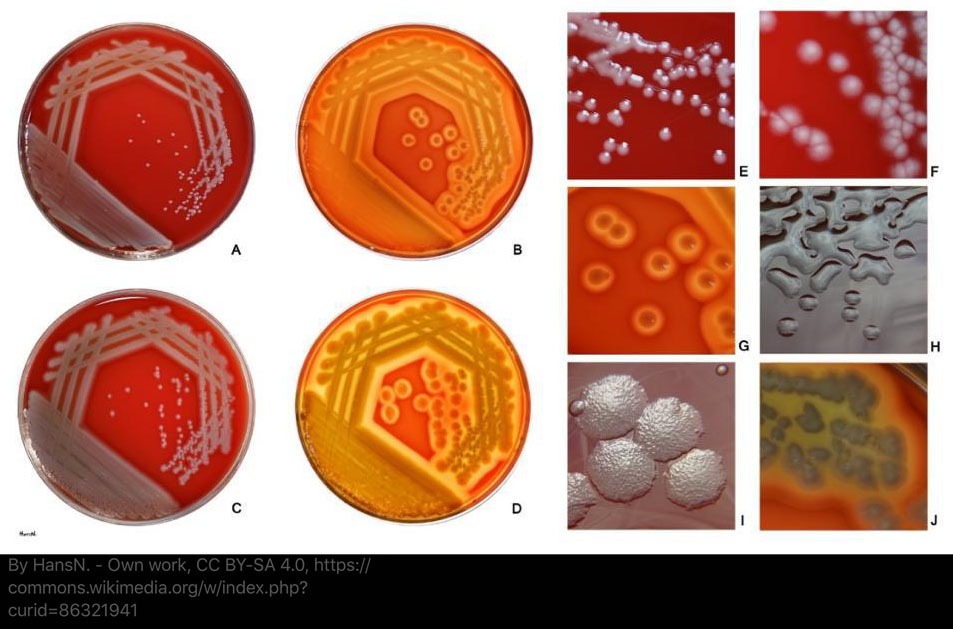
• Entre las bacterias que la OMS califica como prioritarias para investigar por el peligro que representan para la salud humana, se encuentran las Pseudomonas aeruginosa, que se hallan comúnmente en ambientes hospitalarios
• La investigación se realizó en un hospital de Tailandia y extiende la búsqueda de plásmidos como los identificados, y nuevos tipos, en centros hospitalarios de todo el mundo para conocer su prevalencia
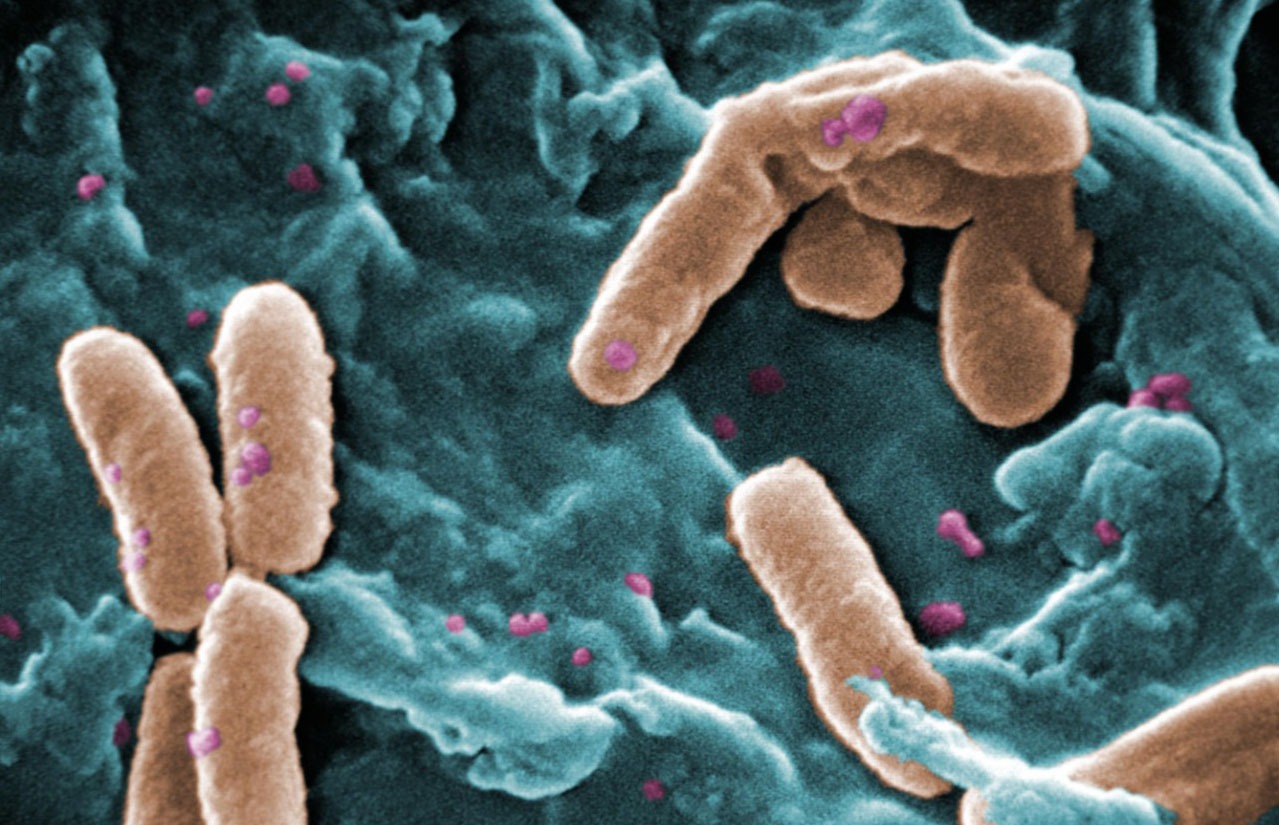
Un nuevo estudio realizado en la Universidad de Liverpool en el Reino Unido, publicado en la revista Nature Communications y co-liderado por un ex-becario postdoctoral de la SECTEI, el doctor Adrián Cazares López, ha revelado una nueva vía por la cual Pseudomonas aeruginosa es capaz de desarrollar resistencia a múltiples antibióticos de relevancia clínica y transferirla a otras bacterias.
El hallazgo es de gran relevancia porque contribuye a dar respuesta al estado de crisis que vive el mundo por el incremento alarmante en los casos de resistencia a antibióticos. El surgimiento de bacterias resistentes al tratamiento ahora amenaza con regresar al planeta a una época oscura en la que cualquier tipo de infección representaba un riesgo mortal.
Son varios los patógenos capaces de causar infecciones resistentes a los fármacos referidos y existe un grupo que la Organización Mundial de la Salud (OMS) ha calificado como prioritario para investigar por el peligro para la especie humana. Entre ellos se encuentra Pseudomonas aeruginosa, que se halla comúnmente en ambientes hospitalarios, donde causa diversos tipos de infecciones que afectan principalmente a personas inmunocomprometidas.
Encontrar nuevas maneras de nulificar bacterias resistentes, especialmente aquellas con resistencia a múltiples fármacos, es tan importante como entender cómo éstas se hicieron resistentes en primera instancia.
El grupo de investigación utilizó tecnologías de secuenciación de vanguardia para descifrar el ADN de bacterias multirresistentes que causaron diferentes tipos de infección en un hospital en Tailandia.
El análisis de la secuencia de ADN condujo a descubrir que varios de los genes que generan la resistencia eran parte de una molécula circular de ADN conocida como plásmido que es, además, flexible. Por ello resulta un vehículo perfecto para transportar genes que ayudan a las bacterias a enfrentar adversidades del ambiente, como los antibióticos.
Un extenso análisis computacional y evolutivo, parte del mismo trabajo, reveló que estos plásmidos no están restringidos a conferir resistencia a las cepas de Tailandia, sino que pueden ser encontrados en otros patógenos de todo el mundo, incluido México. Esta relación ha existido al menos por décadas sin darnos cuenta. Además, el estudio demuestra que la bacteria puede compartir el plásmido con facilidad, en cuestión de horas, convirtiendo en resistentes a bacterias que no lo eran.
El profesor Craig Winstanley, co-líder de la investigación, dijo que “la resistencia a antimicrobianos es un problema global enorme. Si vamos a introducir intervenciones para intentar controlarlo, es esencial que entendamos cómo la resistencia evoluciona y se propaga entre las poblaciones bacterianas.
“Nuestro estudio demuestra la importancia de usar tecnologías de secuenciación de la más alta resolución y análisis de datos complejos que revelen secretos ocultos entre la cantidad masiva de información existente en bases de datos”.
El otro co-líder del estudio, el doctor Cázares López destacó, por su parte, que “las bacterias son organismos increíblemente adaptables que usarán cualquier herramienta a su alcance para prosperar en diversos ambientes, incluyendo las infecciones.
“El trabajo es relevante porque muestra cómo las bacterias pueden encontrar estas herramientas en la naturaleza, en la forma de plásmidos, que les permiten colectar múltiples genes de resistencia y después invadir ambientes clínicos donde causan infecciones mortales. Descubrir estos vínculos en los ambientes naturales y clínicos es primordial para el diseño e implementación de mejores intervenciones antimicrobianas”.
Actualmente, el grupo de investigación extiende la búsqueda de plásmidos como los identificados, y de nuevos tipos, en hospitales de todo el mundo para conocer su prevalencia. Asimismo, intentan descifrar qué hacen a estos plásmidos exitosos en Pseudomonas aeruginosa.
El trabajo fue resultado de una colaboración internacional entre investigadores del Reino Unido, Canadá y Tailandia. La participación del biólogo molecular Adrián Cázares López en el estudio fue posible gracias a la beca que obtuvo de la Secretaría de Educación, Ciencia, Tecnología e Innovación (SECTEI) para cubrir una estancia posdoctoral en la Universidad de Liverpool en 2017.
En la actualidad, el investigador mexicano trabaja en el Instituto Europeo de bioinformática (EMBL-EBI) y el Instituto Wellcome Sanger, asociados a la Universidad de Cambridge en Reino Unido.
La investigación completa lleva por título A megaplasmid family driving dissemination of multidrug resistance in Pseudomonas’ y puede ser consultada desde estas ligas: https://rdcu.be/b20BT o https://www.nature.com/articles/s41467-020-15081-7